本网讯(通讯员 李阳)近日,我校生命科学学院基因组学与抗病遗传科技创新团队取得重要研究成果。
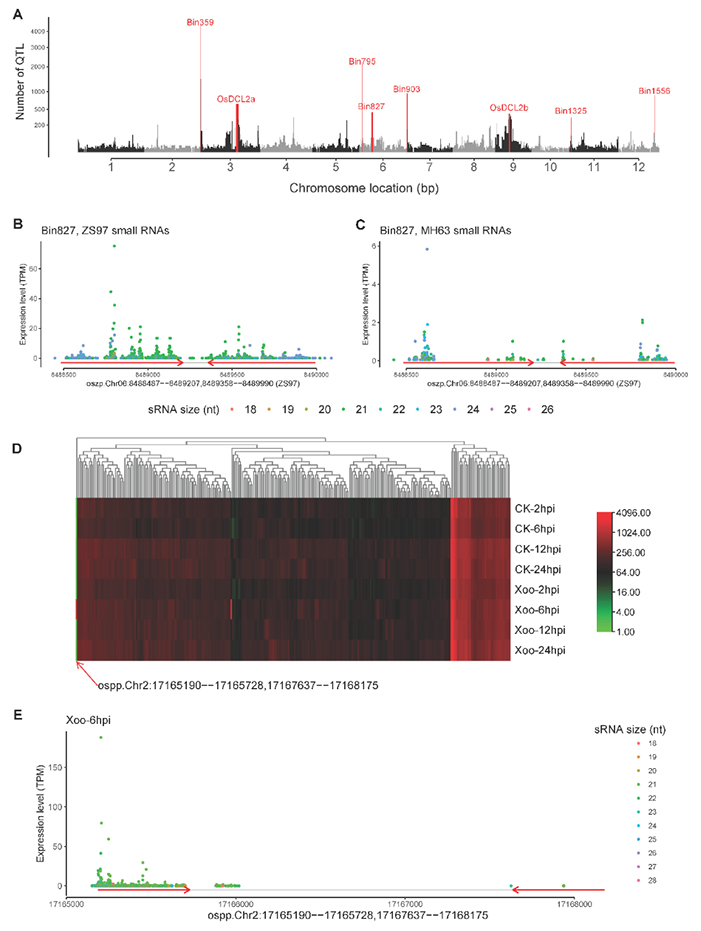
小干扰RNA(small interfering RNA,siRNA)是一类在真核生物中广泛存在、长度为18~25个碱基的非编码调控序列。siRNA通过调节基因的表达水平,参与了真核生物的生长发育、肿瘤发生、性别分化、环境适应等众多生命过程。研究发现,LIR可转录形成长的发卡结构RNA(long hairpin RNA, hpRNA),经进一步加工可形成长度为21~22个碱基的siRNA。
鉴于LIR的重要生物学功能,姚文教授课题组联合中国科学院北京基因组研究所系统鉴定了424个真核生物基因组中的6,619,473条LIR序列,并构建了LIRBase数据库。其中,在77个后生动物基因组中鉴定了297,317条LIR序列;在139个植物基因组鉴定了1,731,978条LIR序列;在208个脊椎动物基因组中鉴定了4,590,178条LIR序列。作为一站式数据库,LIRBase将有助于推动真核生物LIR以及由LIR形成的siRNA的功能及进化机制研究。
该成果以“LIRBase: a comprehensive database of long inverted repeats in eukaryotic genomes”为题在线发表于生物大类一区Top期刊Nucleic Acids Research。我校博士生贾利华和校聘副教授李阳为该论文共同第一作者,姚文和中国科学院北京基因组研究所章张研究员为该论文共同通讯作者。该研究得到了国家重点研发计划、国家自然科学基金、河南农业大学拔尖人才科研启动基金、中国科学院战略性先导科技专项等项目的资助。
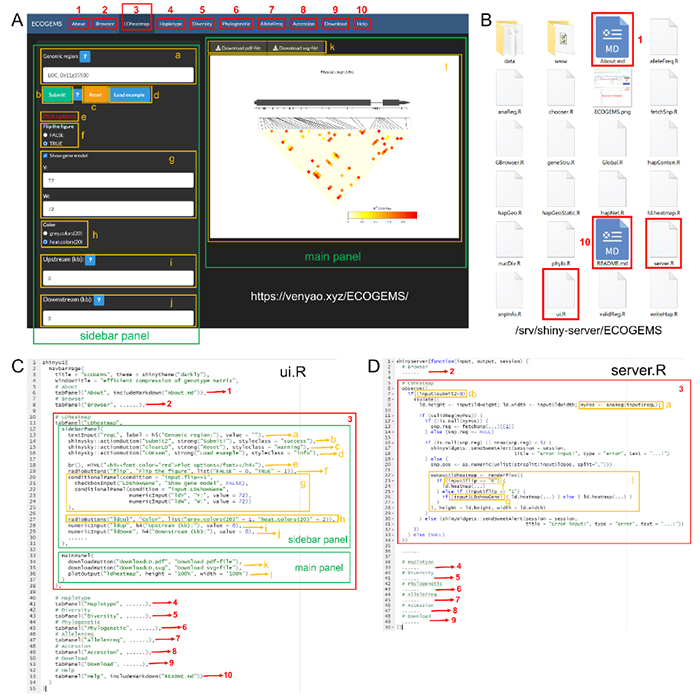
R/Shiny可用于快速搭建动态网络应用程序进行数据分析和可视化,将极大促进交互式网络应用程序在生物数据分析领域中的应用。
姚文和张会勇教授课题组调研了不同计算机编程语言在生物学网络应用程序构建中的应用,总结了基于R/Shiny构建生物学网络应用程序的最新进展,介绍了使用R/Shiny构建生物学网络应用程序的基本框架和流程,总结了使用R/Shiny构建生物学网络应用程序的一些要点和注意事项,同时评估了R/Shiny在构建生物学网络应用程序中的优势及不足之处。
该成果以“Development of interactive biological web applications with R/Shiny”为题在生物大类Top期刊Briefings in Bioinformatics在线发表。我校博士生贾利华为该论文第一作者,姚文和张会勇为该论文共同通讯作者。该研究得到了国家自然科学基金、河南农业大学拔尖人才科研启动基金、河南省科技攻关项目的资助。
【全文链接】
1.https://academic.oup.com/nar/advance-article/doi/10.1093/nar/gkab912/6395344
2.https://academic.oup.com/bib/advance-article-abstract/doi/10.1093/bib/bbab415/6387320?redirectedFrom=fulltext
编辑/杨钰晨 签审/张朝阳 审核/谢东明